各位老师,大家好!经过上一篇《数据库挖掘实操 | (一)CMap——超简单易上手的潜在药物数据库》(点击标题查看详情)的学习,我们了解掌握了一个简单易上手的药物数据库CMap的具体分析实操。今天我们步履不停,继续为大家介绍一个新的免疫组化验证数据库——HPA数据库,帮助大家实现不做实验也能得到的免疫组化验证结果图像,我们一起来看一看吧~
♦HPA数据库介绍
人类蛋白质图谱(The Human Protein Atlas,简称HPA,https://www.proteinatlas.org/)由瑞典Knut & Alice Wallenberg基金会于2003年创建。
HPA基于蛋白组学、转录组学以及系统生物学等多种组学技术,绘制组织、细胞、器官中的所有蛋白质图谱。HPA提供全部24,000多种人类蛋白质的组织和细胞分布信息,提供了每一种蛋白质在多种人类正常组织、肿瘤组织、细胞系和血液细胞内的分布和表达情况。大部分结果还带有用免疫组化染色图、免疫荧光共聚焦照片等图片信息,并有专业人员标引表达水平,可以直接应用于文献展示。
资源中的所有数据都开放访问,目前这个网站只针对人类蛋白质。
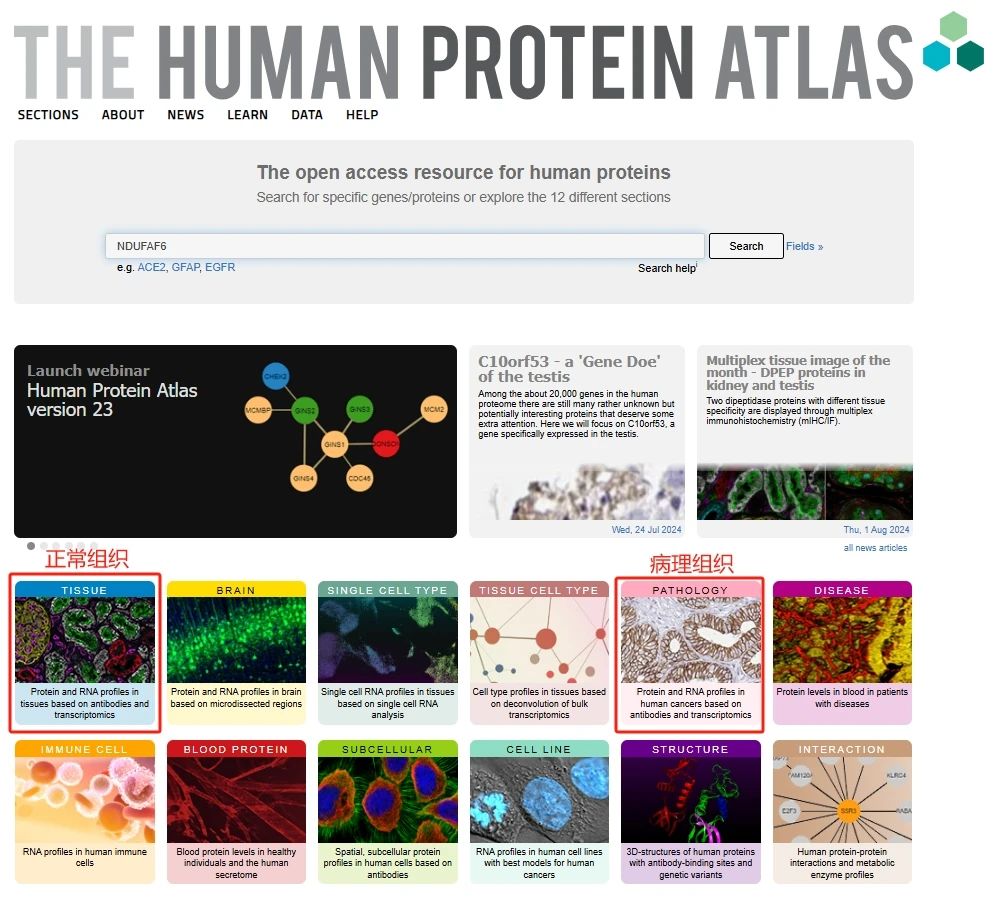 图1:HPA网站首页
图1:HPA网站首页
人类蛋白质图谱HPA数据库由十二个独立的部分组成,每个部分侧重于人类蛋白质全基因组分析的特定方面。今天的文章主要是基于其中两个部分——组织tissue、病理pathology图谱,详细教学如何在HPA数据库中找到特定蛋白在正常组织和特定癌症组织中的免疫组化图片,真正实现不做实验也能获取免疫组化验证结果!话不多说,我们直接进入实操教学!
♦HPA实操教学
我们以一篇研究乳腺癌的文章[1]作为案例,进行实操的详细讲解。在该案例中,作者通过HPA数据库中的NDUFAF6的免疫组化结果,验证了NDUFAF6在正常组织(上)与乳腺癌肿瘤(下)中的表达差异,即肿瘤组织中NDUFAF6的表达水平比正常组织中更高。
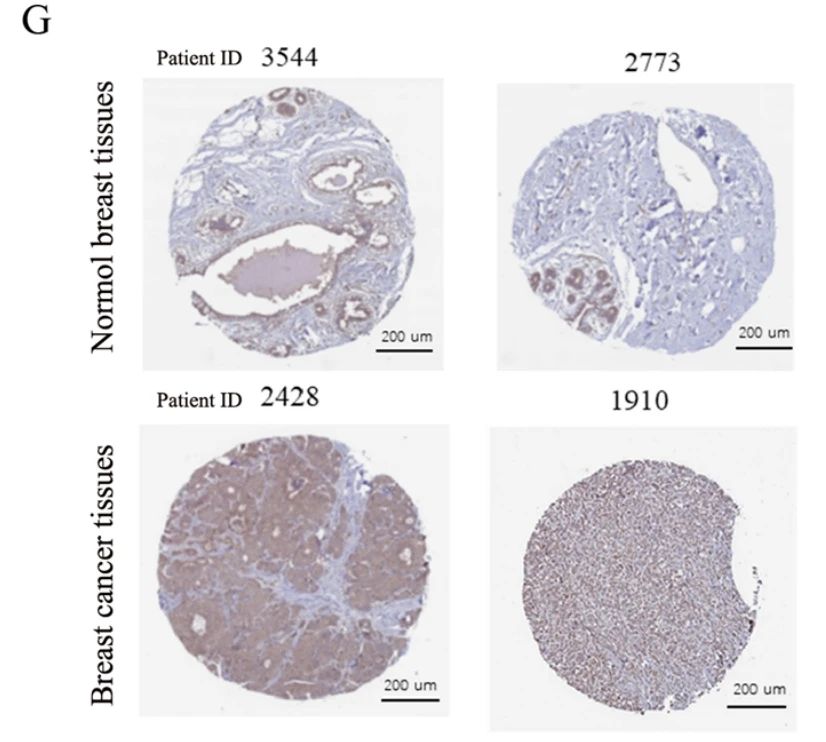 图2:NDUFAF6蛋白的HPA数据库结果
图2:NDUFAF6蛋白的HPA数据库结果
(图片摘自文献Impact of NDUFAF6 on breast cancer prognosis: linking mitochondrial regulation to immune response and PD-L1 expression[1]:展示从HPA 数据库获取的正常乳腺组织和乳腺癌肿瘤组织中NDUFAF6的免疫组化结果。)
以下是使用HPA数据库,并从中获得目标免疫组化图片的具体操作步骤:
1.目标检索:
首先打开HPA网站(https://www.proteinatlas.org/),在搜索栏输入目标基因或蛋白名称,如本例中的“NDUFAF6”,而后点击“Search”。
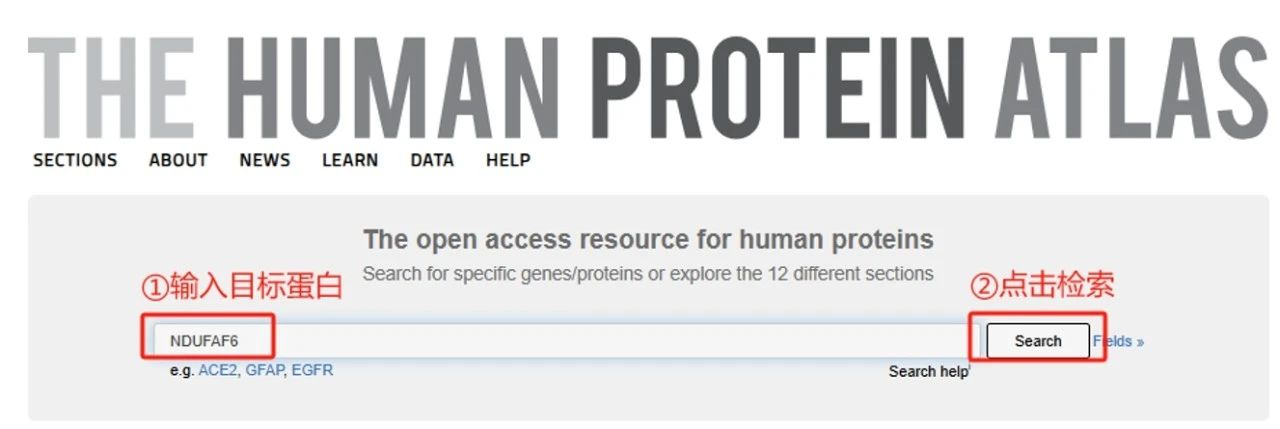 图3:HPA数据库首页检索
图3:HPA数据库首页检索
页面跳转至搜索结果,包括对该基因或蛋白的描述,以及各种图谱结果(Tissue、Brain、Single cell、Pathology、Disease、Immune、Blood、Subcell、Cell line、Structure、Interaction)。
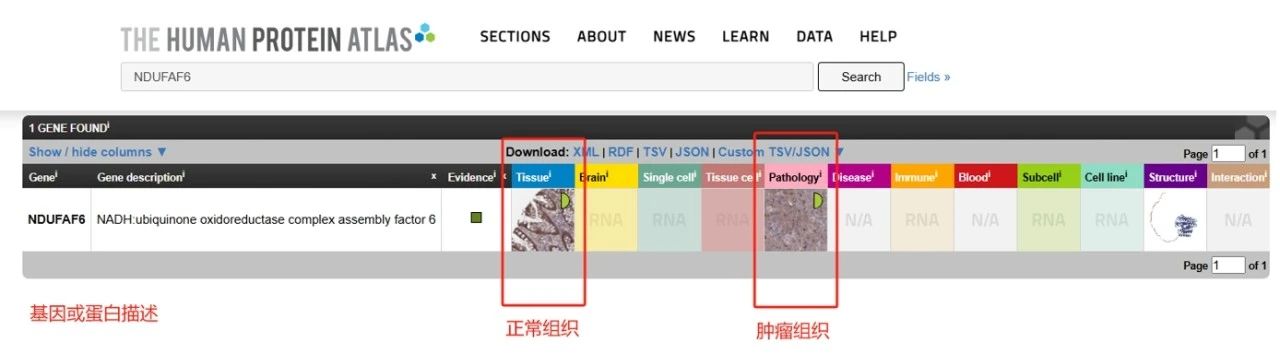 图4:正常组织及病理组织等12种图谱页面
图4:正常组织及病理组织等12种图谱页面
2.获取
正常组织免疫组化图片:首先点击“Tissue”栏,跳转后,该页面主要包括一般信息(基因名称、描述、蛋白分类、预测位置、转录本数目),蛋白图谱信息(RNA表达组织特异性、RNA表达组织分布、蛋白证据等级、蛋白表达定位)和免疫组化数据情况(数据可靠性、评分、相应抗体)。
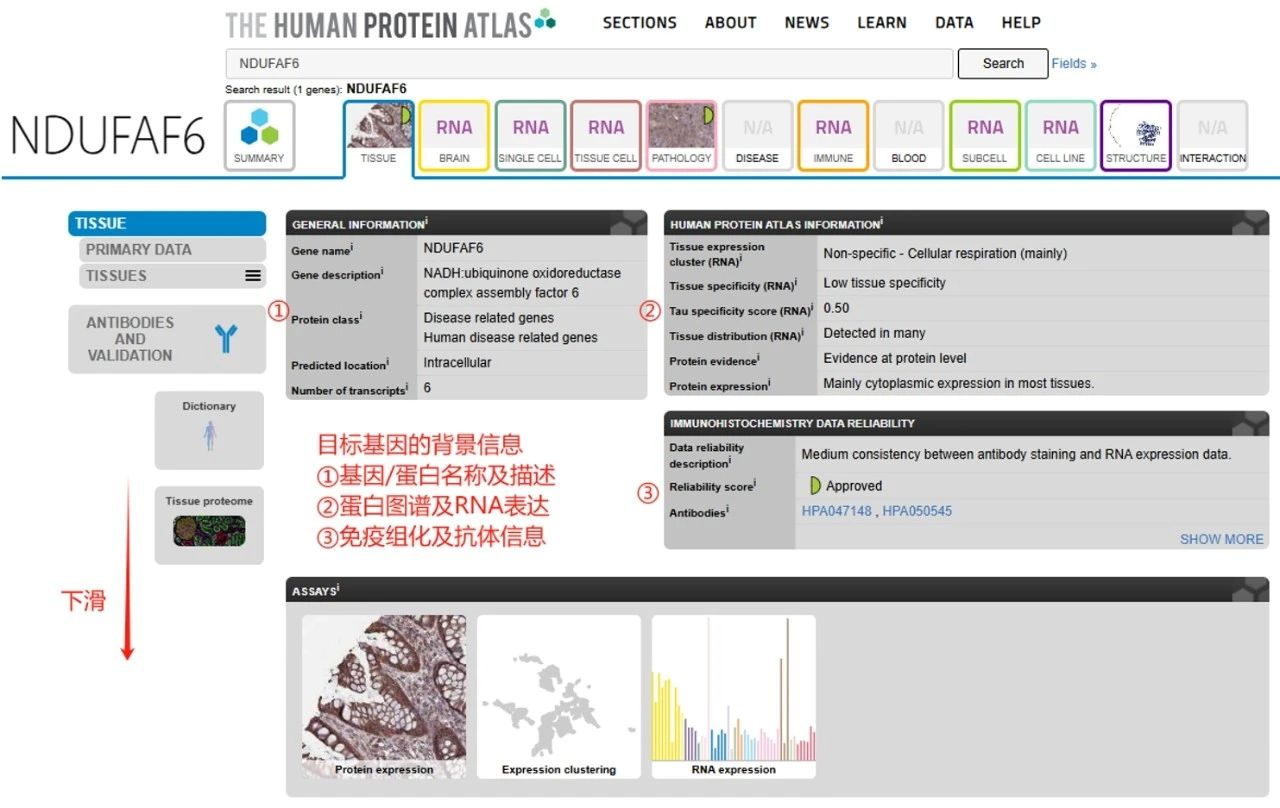 图5:正常组织详细页面
图5:正常组织详细页面
下拉页面到达RNA和蛋白表达图谱,按照人体器官分布,左侧显示的是RNA表达,右侧是蛋白的表达。共展示了44种正常人体组织的蛋白质表达数据,涵盖了15323个基因/蛋白。可以看到基因/蛋白在不同系统中的器官中的表达情况。
本例中研究的疾病是乳腺癌,因此我们选择对应的女性器官中的乳腺组织 Breast,点击进一步获取免疫组化结果。
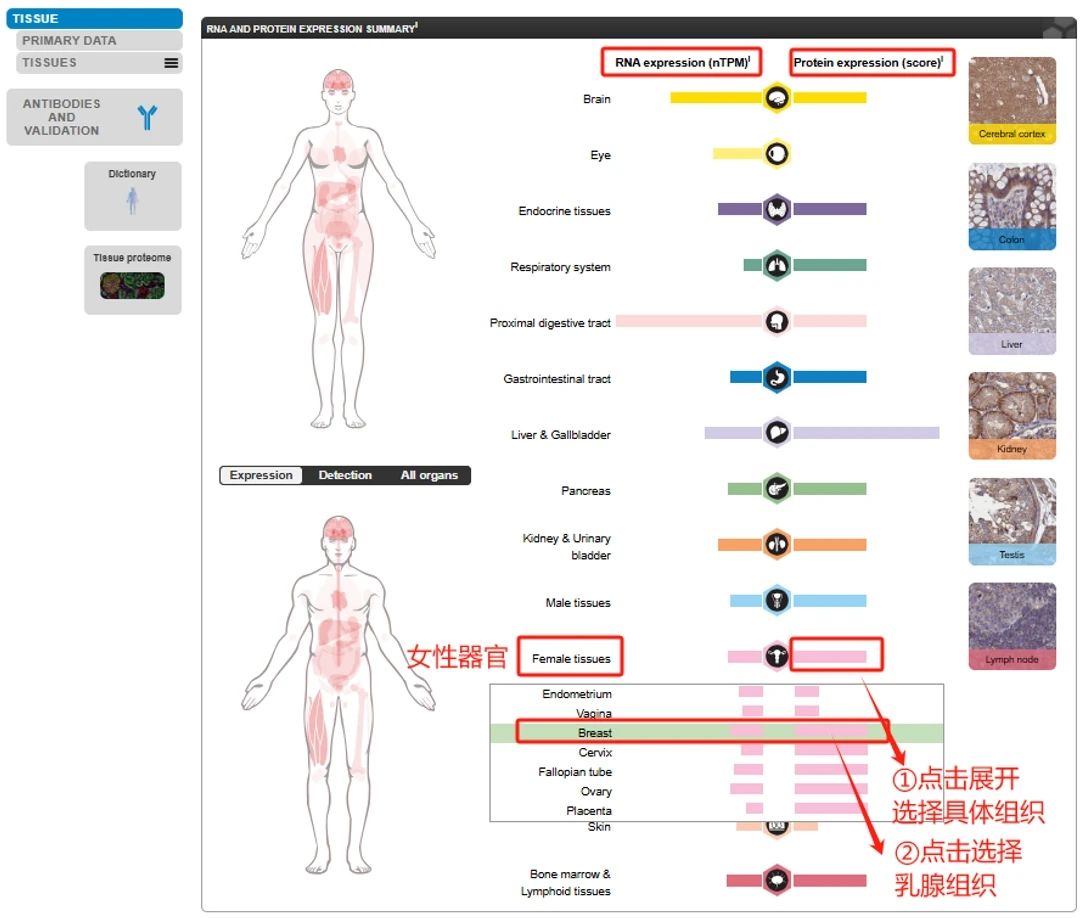 图6:RNA及蛋白质表达页面
图6:RNA及蛋白质表达页面
进入具体的组织免疫组化页面,选择合适的免疫组化图片。这里需要注意的是:我们需要记录抗体编号HPA047148这一数据,在后续选择肿瘤组织的免疫组化结果时,将根据该信息选择同一抗体下的免疫组化结果。之后根据自己的分析情况,挑选合适的免疫组化结果。文章中选择的免疫组化图片就是图7中圈选的两张,点击图片可进一步查看详细信息,并进行原图的下载。
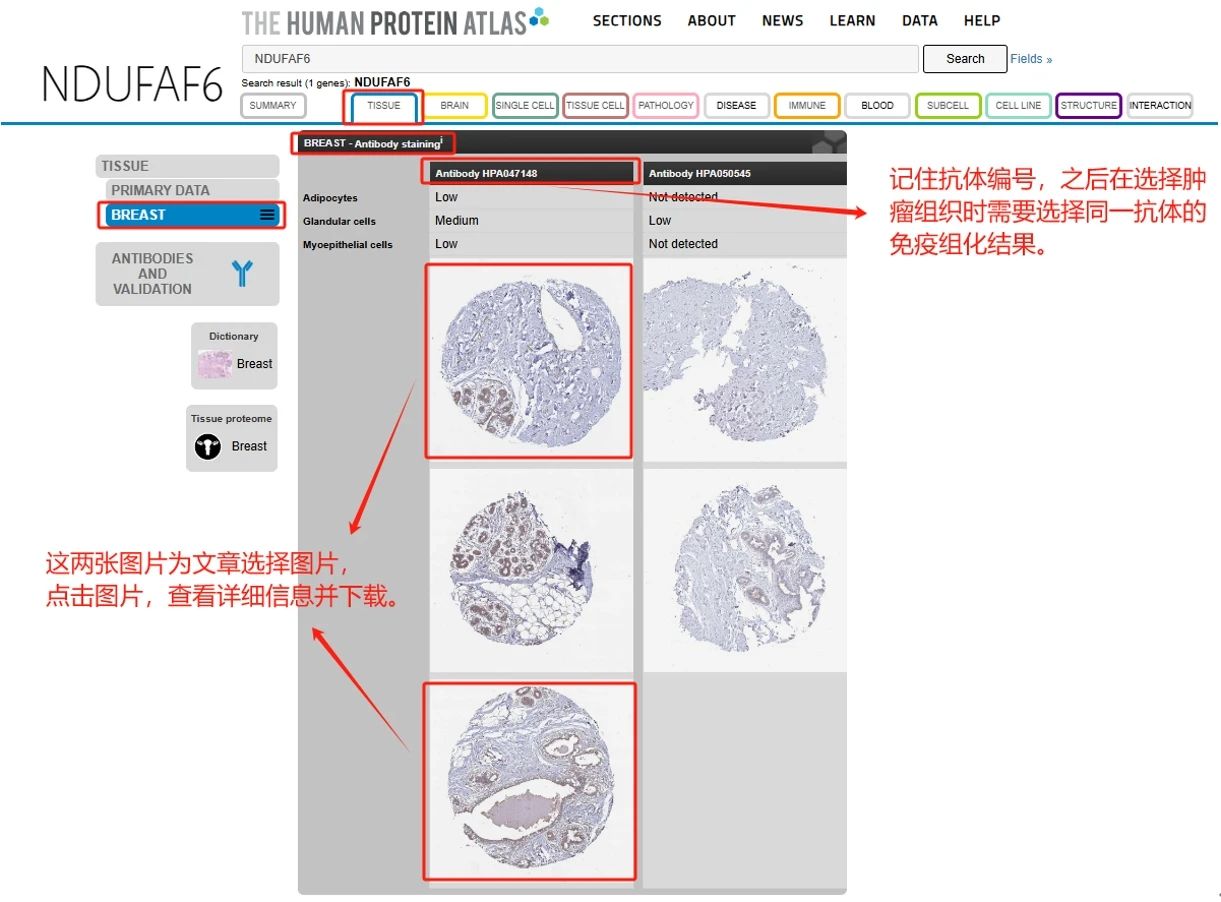 图7:胸腺正常组织免疫组化页面
图7:胸腺正常组织免疫组化页面
3.获取正常组织免疫组化图片:
回到图3搜索页面,点击“Pathology”栏,下滑至最底部,寻找肿瘤组织中的免疫组化图片,HPA涵盖了目前常见的17种人类癌症类型。
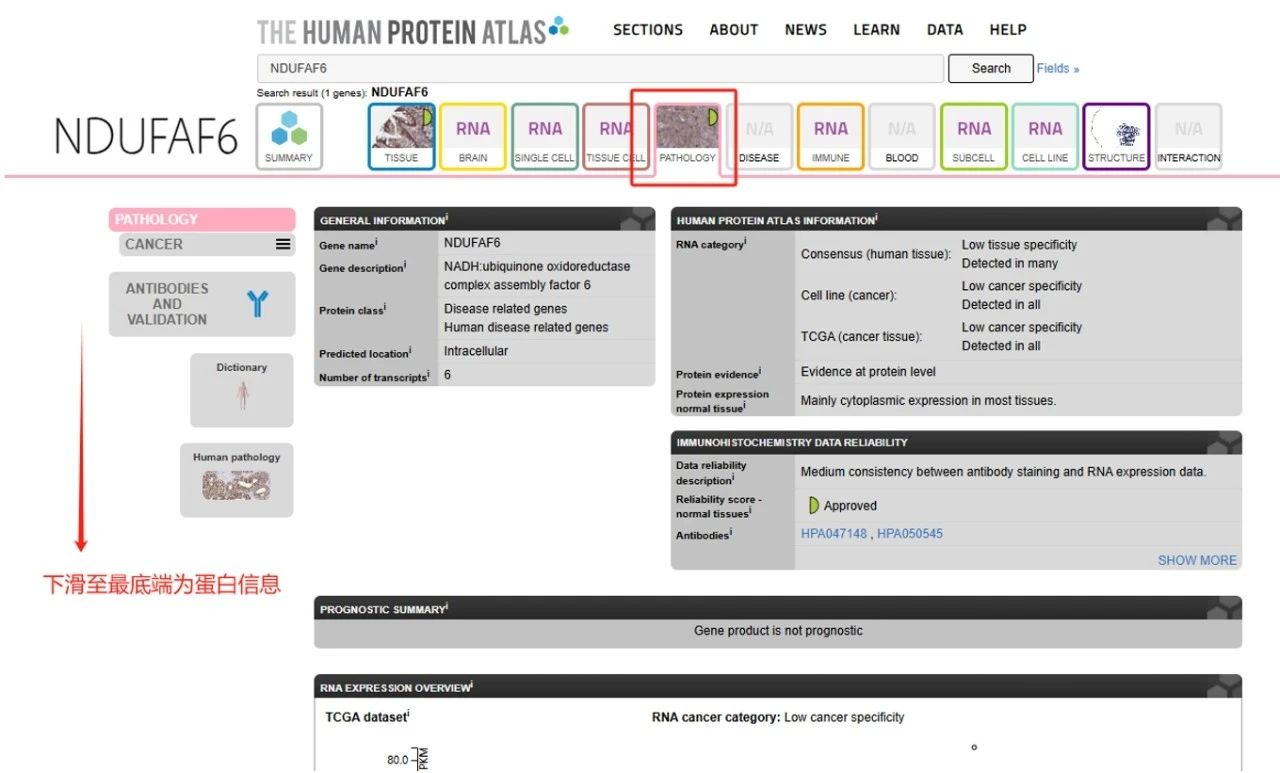 图8:肿瘤组织详细页面
图8:肿瘤组织详细页面
此时抗体选择与正常组织保持一致,根据研究内容,此处选择乳腺癌“Breast Cancer”。
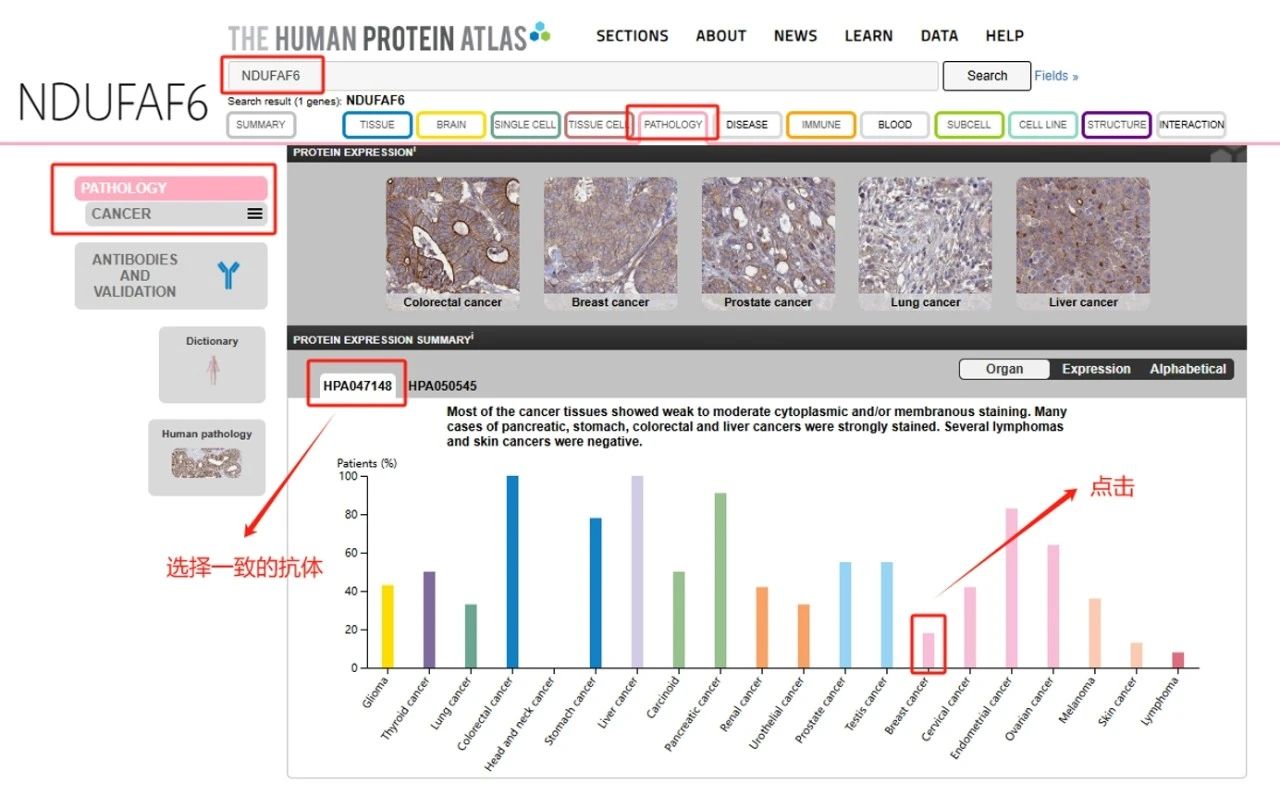 图9:不同肿瘤组织分类页面
图9:不同肿瘤组织分类页面
进入具体的肿瘤组织免疫组化页面,根据自己的分析情况,选择合适的免疫组化图片,文章中选择的免疫组化图片就是图10中圈选的两张。
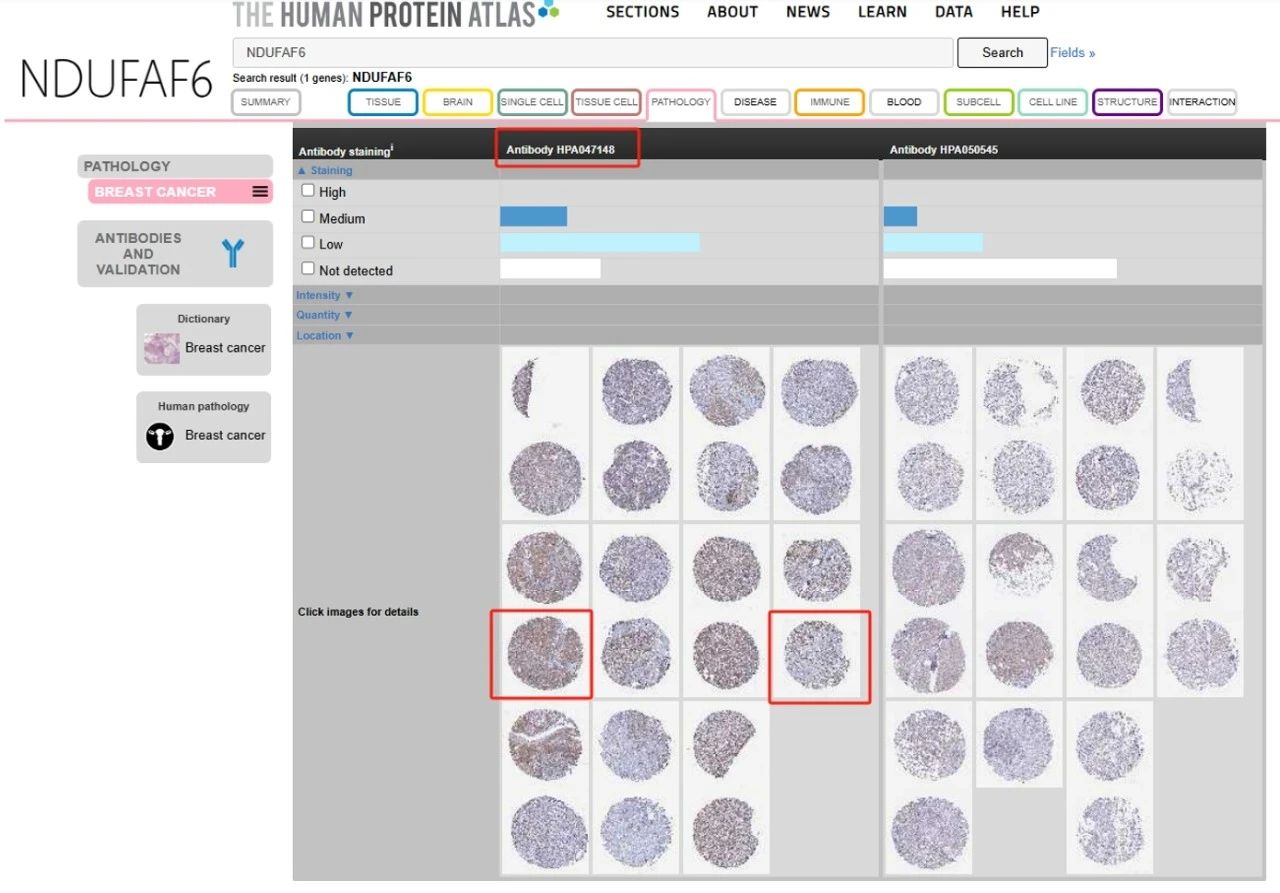 图10:乳腺癌肿瘤组织免疫组化页面
图10:乳腺癌肿瘤组织免疫组化页面
到这里,如何从HPA数据库挑选目标蛋白的免疫组化图片步骤就全部结束了!我们通过具体的案例体会了HPA数据库的应用价值——挑选到的免疫组化图片可以作为验证结果直接放在文章中,这对于缺少验证的文章绝对是一大助力,论文的档次和完整性将大大得到提升~
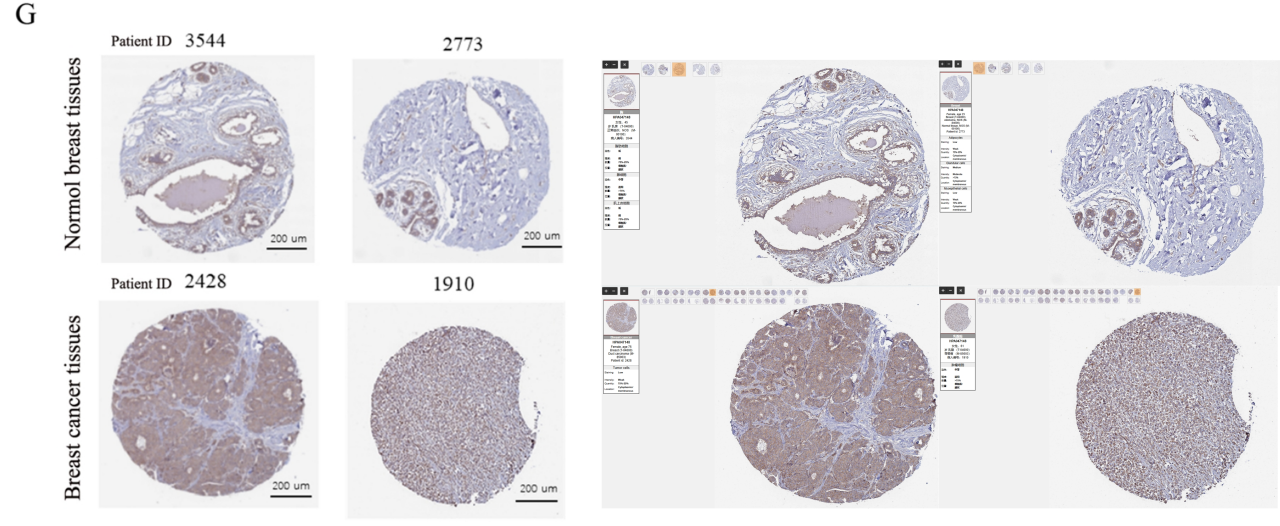 图11:文章免疫组化结果(左)与挑选免疫组化结果(右)对比
图11:文章免疫组化结果(左)与挑选免疫组化结果(右)对比
【挑选步骤】简化版:
从HPA数据库中获取到免疫组化结果:
进入HPA数据库(https://www.proteinatlas.org/)→输入基因名称(NDUFAF6)→点击“Search” →选择“Tissue“→选择”breast“→挑选正常组织免疫组化图片→回到第二张图的界面选择”Pathology“→选择”breast cancer“→挑选病理组织免疫组化图片。
♦论文使用HPA图片的文字表述
获取了HPA免疫组化图片后,在论文写作中该如何描述呢?下面分享两篇文献中关于HPA数据库的应用描述:
文献1. Impact of NDUFAF6 on breast cancer prognosis: linking mitochondrial regulation to immune response and PD-L1 expression.[1]

 图12:HPA结果部分的应用描述(上)与方法学描述(下)
图12:HPA结果部分的应用描述(上)与方法学描述(下)
HPA结果部分应用描述(上):“为了证实NDUFAF6的蛋白水平在乳腺癌中的表达高于正常乳腺组织,我们分析了HPA数据库的免疫组化结果并收集临床样本进行免疫组化实验。结果表明,乳腺癌组织中的蛋白表达水平明显高于正常乳腺组织。IHC的典型显微照片如图1G、Hh所示。研究清楚地证明了NDUFAF6在乳腺癌组织中的显著过表达,为其作为乳腺癌治疗靶点的潜力提供了强有力的证据。”
HPA方法学描述(下):“利用人类蛋白图谱(Human Protein Atlas, HPA)数据库和免疫组化技术,进一步评估了NDUFAF6在组织样本中的局部表达模式。”
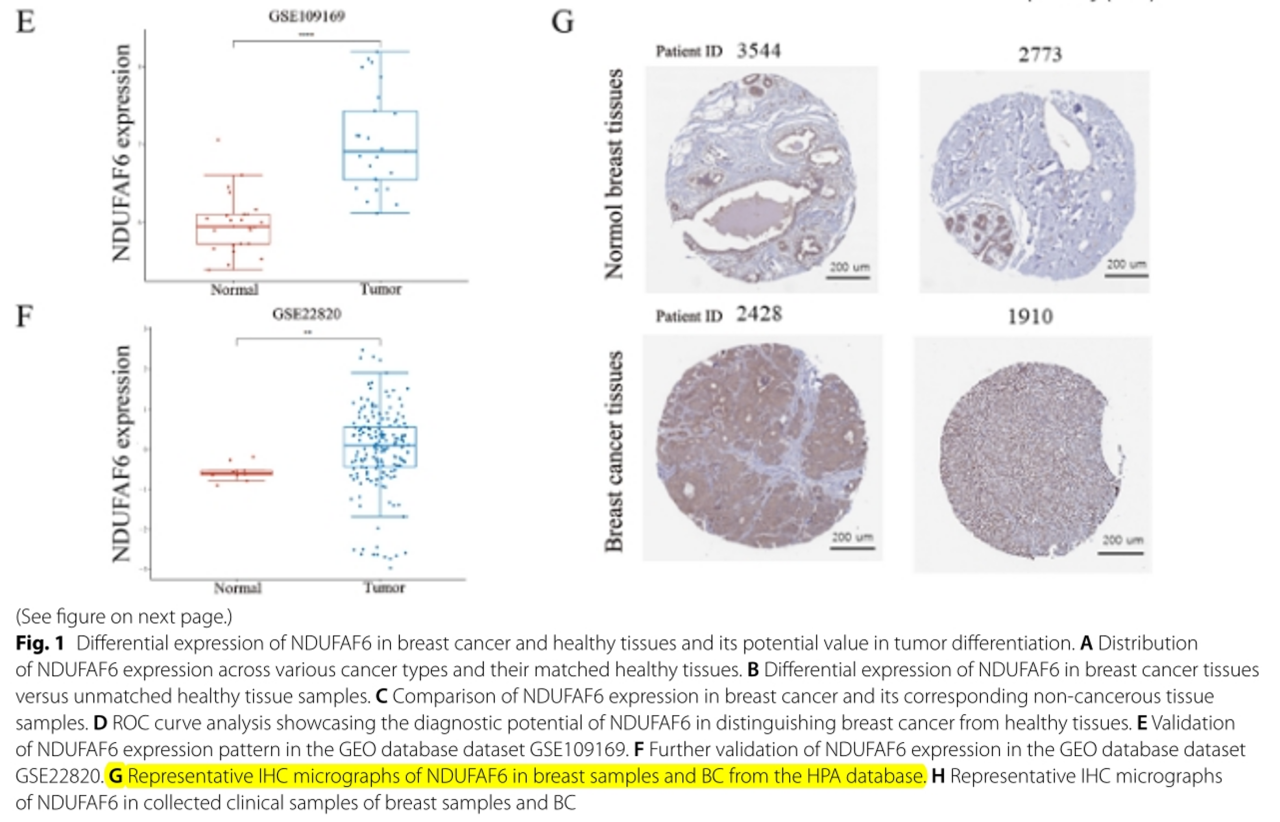 图13:HPA主图展示免疫组化结果
图13:HPA主图展示免疫组化结果
文献2. Identification of plasma proteomic signatures associated with the progression of cardia gastric cancer and precancerous lesions.[2]

 图14:HPA结果部分的应用描述(上)与方法学描述(下)
图14:HPA结果部分的应用描述(上)与方法学描述(下)
HPA结果部分应用描述(上):“我们使用来自HPA数据库的免疫组织化学结果进一步验证了五种候选蛋白生物标志物在胃癌和正常组织之间的表达(图E) 。……结合HPA公共数据库免疫组化图像的定量信息,进一步验证了候选蛋白在正常组织和胃癌组织中的表达水平,与DIA-MS结果一致。”
HPA方法学描述(下):“此外,还使用Human Protein Atlas (HPA) (http://www.proteinatlas.org/)来确认验证蛋白的表达。”
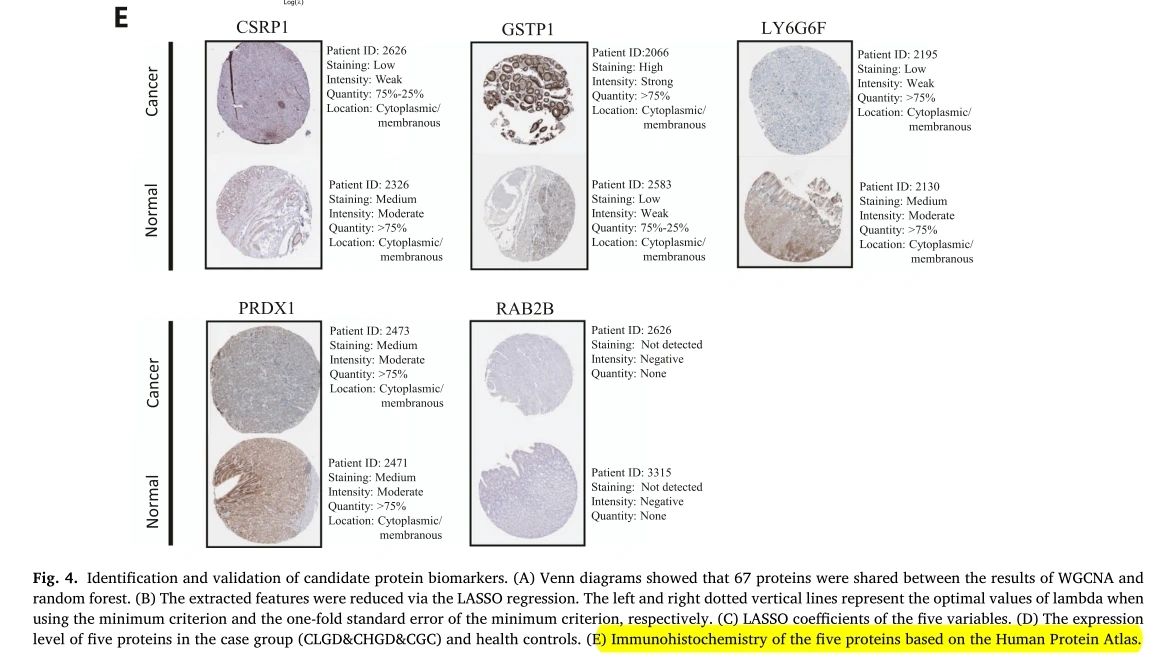 图15:HPA主图展示免疫组化结果
图15:HPA主图展示免疫组化结果
♦“黄金搭档”——蛋白质组学+HPA数据库
蛋白质组学通过高通量无偏性的检测,可以获得大量蛋白的表达数据,并通过生信分析找出与生理病理相关的关键蛋白质;而HPA数据库收录了大量不同组织中蛋白的生理病理免疫组化结果,是对组学数据的绝佳验证和补充。二者可谓是“黄金搭档”,因此可以采用蛋白质组学-生信/统计分析-HPA验证这个流程(图16),无论研究者是否针对目标蛋白开展免疫组化验证,都可以在自己的论文中补充公开数据库的免疫组化验证结果,为研究的可靠性提供数据支持。
 图16:HPA应用于第三步生物标志物的验证
图16:HPA应用于第三步生物标志物的验证
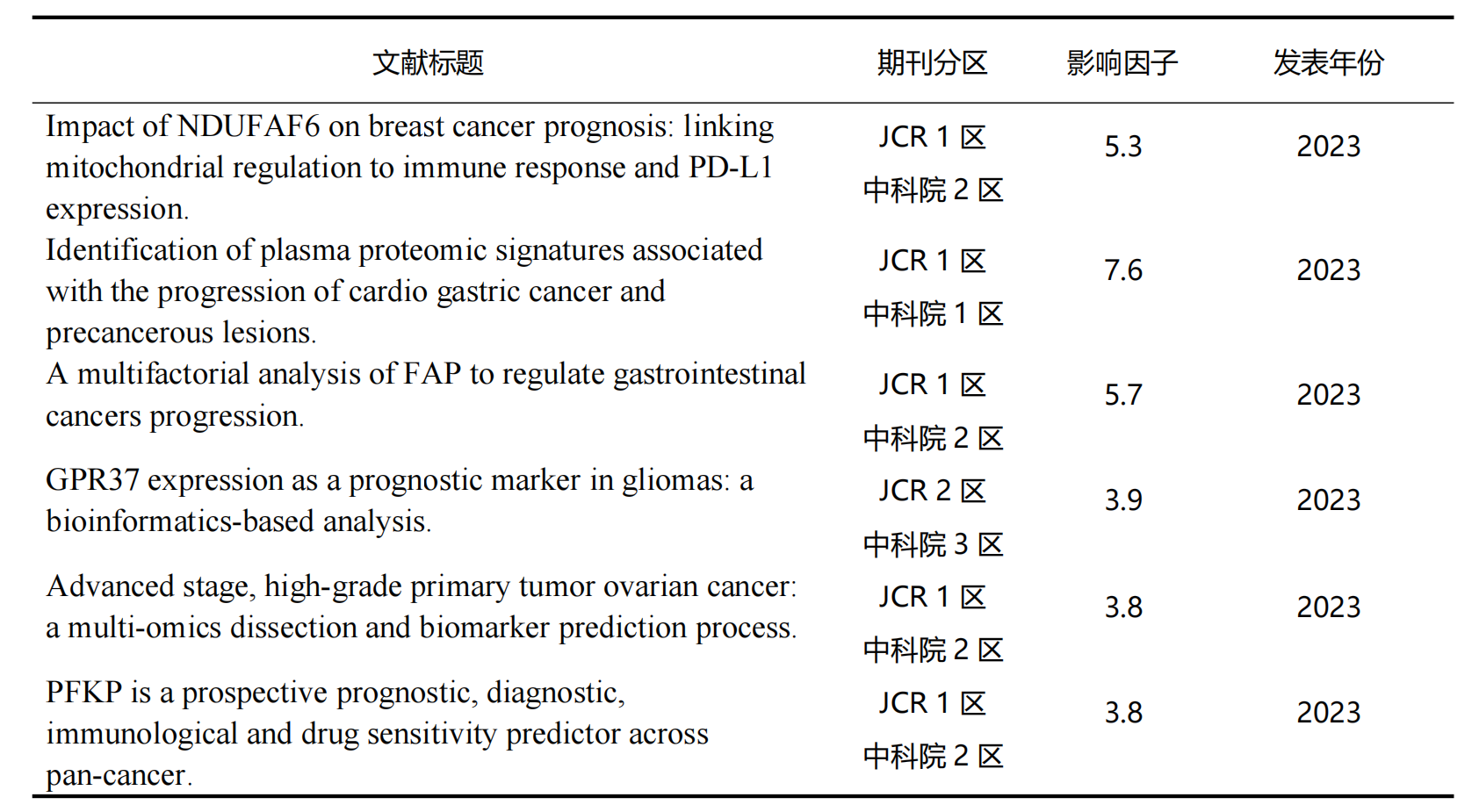
表1:挖掘HPA免疫组化验证结果的文献举例[1-5]
如果您还是觉得一个一个蛋白搜索太麻烦,那就使用我们的FFPE样本蛋白质组研究一站式解决方案吧!HPA数据库分析结果已经整合到我们的报告中,提供目标蛋白HPA免疫组化检索列表,链接定位到目标蛋白免疫组化结果网址,可直接保存网站图片增添至文章结果中。为您大大节约时间、减少工作量!
参考文献
[1]Jiang B, Wu S, Zeng L, et al. Impact of NDUFAF6 on breast cancer prognosis: linking mitochondrial regulation to immune response and PD-L1 expression[J]. Cancer Cell Int, 2024,24(1):99.
[2]Gu J, Xie S, Li X, et al. Identification of plasma proteomic signatures associated with the progression of cardia gastric cancer and precancerous lesions[J]. J Natl Cancer Cent, 2023,3(4):286-294.
[3]Liang K, Guo Z, Zhang S, et al. GPR37 expression as a prognostic marker in gliomas: a bioinformatics-based analysis[J]. Aging (Albany NY), 2023,15(19):10146-10167.
[4]Honar Y S, Javaher S, Soleimani M, et al. Advanced stage, high-grade primary tumor ovarian cancer: a multi-omics dissection and biomarker prediction process[J]. Sci Rep, 2023,13(1):17265.
[5]Peng J, Li P, Li Y, et al. PFKP is a prospective prognostic, diagnostic, immunological and drug sensitivity predictor across pan-cancer[J]. Sci Rep, 2023,13(1):17399.


















