期刊名称:Microbiome
影响因子:13.8
期刊分区:JCR 1区/生物学 1区
发表时间:2024年1月
作者单位:浙江省肿瘤医院、西湖大学医学院
相关疾病:胃癌
样本类型:舌苔样本
样本数量:417
其他信息:宏蛋白质组学、机器学习模型、生物标志物、多中心队列
♦前言
今天与大家分享一篇利用蛋白质组学技术检测胃癌患者的舌苔样本,以揭示胃癌患者口腔环境中微生物与宿主之间相互作用的文章。研究团队基于舌苔微生物蛋白,建立了胃癌筛查模型,为胃癌的早期诊断提供了新的可能性。该文章于2024年1月发表在《Microbiome》(IF=13.8)杂志上,标题为“In-depth metaproteomics analysis of tongue coating for gastric cancer: a multicenter diagnostic research study”。

♦研究背景
胃癌是全球常见的消化系统恶性肿瘤之一,在中国的发病率和死亡率均居高不下。早期胃癌往往没有明显的症状,很多患者在发现时已经处于进展期。内窥镜检查是目前胃癌诊断使用的最可靠的侵入性筛查方法,但需要配备专业的设备和经验丰富的医疗人员,并且患者的体验欠佳。为了改善广泛采用内窥镜检查受到阻碍的现状,我们迫切需要一种既精准又广泛适用的方法来识别高风险的胃癌个体。
人体有五大微生态系统,包括消化道、呼吸道、泌尿生殖道、口腔和皮肤。其中,口腔内存在数百到数千种细菌,口腔微生物稳态的失衡不仅可能导致口腔疾病,还可能影响全身健康,例如增加恶性肿瘤的风险。舌苔作为舌背上覆盖的一层物质,由上皮细胞、代谢物和微生物等组成,既是口腔微生物的栖息地,又与消化道直接相连,因此舌苔可以反映胃部的健康状况。
早在两千多年前,中医就已经提出了“舌诊”理论,而现代研究者也在探索舌头所隐含的健康信息。目前,大多数相关研究是基于宏基因组学对舌苔特征进行描述,而随着宏蛋白质组学的发展,研究者们开始在蛋白质水平分析微生物在疾病中的作用。目前,口腔宏蛋白质组学的研究主要集中在牙龈龋齿或口腔恶性肿瘤上,对胃癌患者的口腔宏蛋白质组学研究尚未展开。
因此,本文旨在通过对胃癌患者和非胃癌患者的舌苔样本进行宏蛋白质组学检测,探索一种新的非侵入性胃癌筛查方法。
♦研究样本
研究共收集了来自5个独立研究中心的180名胃癌患者和185名非胃癌患者的417份舌苔样本。
其中由来自某实验室的5名健康个体的57份样本构成时间队列。该队列用于研究舌苔蛋白质组随时间变化的稳定性。来自浙江省肿瘤医院120例胃癌患者和120例非胃癌患者的舌苔样本,组成发现队列。来自另外三家医院的60名胃癌患者和60名非癌患者的舌苔样本组成多中心队列,作为外部验证队列。

图1 研究样本组成情况
♦实验设计
研究首先利用时间队列的舌苔样本数据,发现随着时间推移,舌苔中的人源蛋白相对稳定,而微生物来源蛋白在不同个体间稳定性较低。这一发现为后续分析奠定了基础。接下来,研究对鉴定到的人源蛋白和微生物来源蛋白进行了功能注释和富集分析,以揭示这些蛋白在胃癌患者中的潜在生物学作用,从而更好地理解它们在疾病中的角色。
随后,研究重点分析了舌苔微生物群的分类,并探讨了舌苔微生物与胃癌发生之间的关联。考虑到舌苔微生物与宿主之间可能存在相互影响,研究进一步筛选了人源蛋白中的差异表达蛋白,并探讨了这些差异表达蛋白与微生物来源蛋白的潜在关系,以揭示胃癌患者口腔环境中微生物与宿主之间的相互作用。
最后,鉴于微生物来源蛋白在个体间具有差异性,研究尝试基于这些蛋白建立了胃癌筛查模型,以探索一种新的非侵入性胃癌筛查方法。

图2 研究流程
♦实验结果
(1)构建舌苔蛋白序列库
由于地域、饮食和健康状况的差异,现有的公共蛋白数据库难以充分反映人类舌苔样本的蛋白质组特征。因此研究首先从健康个体、胃炎患者、I-II期胃癌患者以及III-IV期胃癌患者中各随机抽取了20份舌苔样本,从每个样本中提取了等量DNA,并将同一组的DNA样本混合后进行宏基因组测序,构建了微生物来源蛋白数据库。随后由于舌苔中既包含人源蛋白也有微生物来源蛋白质,研究便将微生物来源蛋白数据与UniProt数据库中的人源蛋白数据整合,构建了特殊的舌苔蛋白序列库(图3B)。
由于舌苔样本具有特殊性,接下来研究使用了一种压力循环技术(PCT),该技术通过常压和超高压的交替循环,能有效破碎细胞并提取蛋白质等重要生物分子。最后将舌苔样本中提取出的蛋白酶解成肽段后进行上机检测,先基于DDA定量检测数据建立了针对舌苔样本的特殊谱库,随后将样本的DIA定量检测数据依托于这一特殊谱库进行搜库鉴定,对舌苔样本中的蛋白进行识别和定量(图3C-D)。
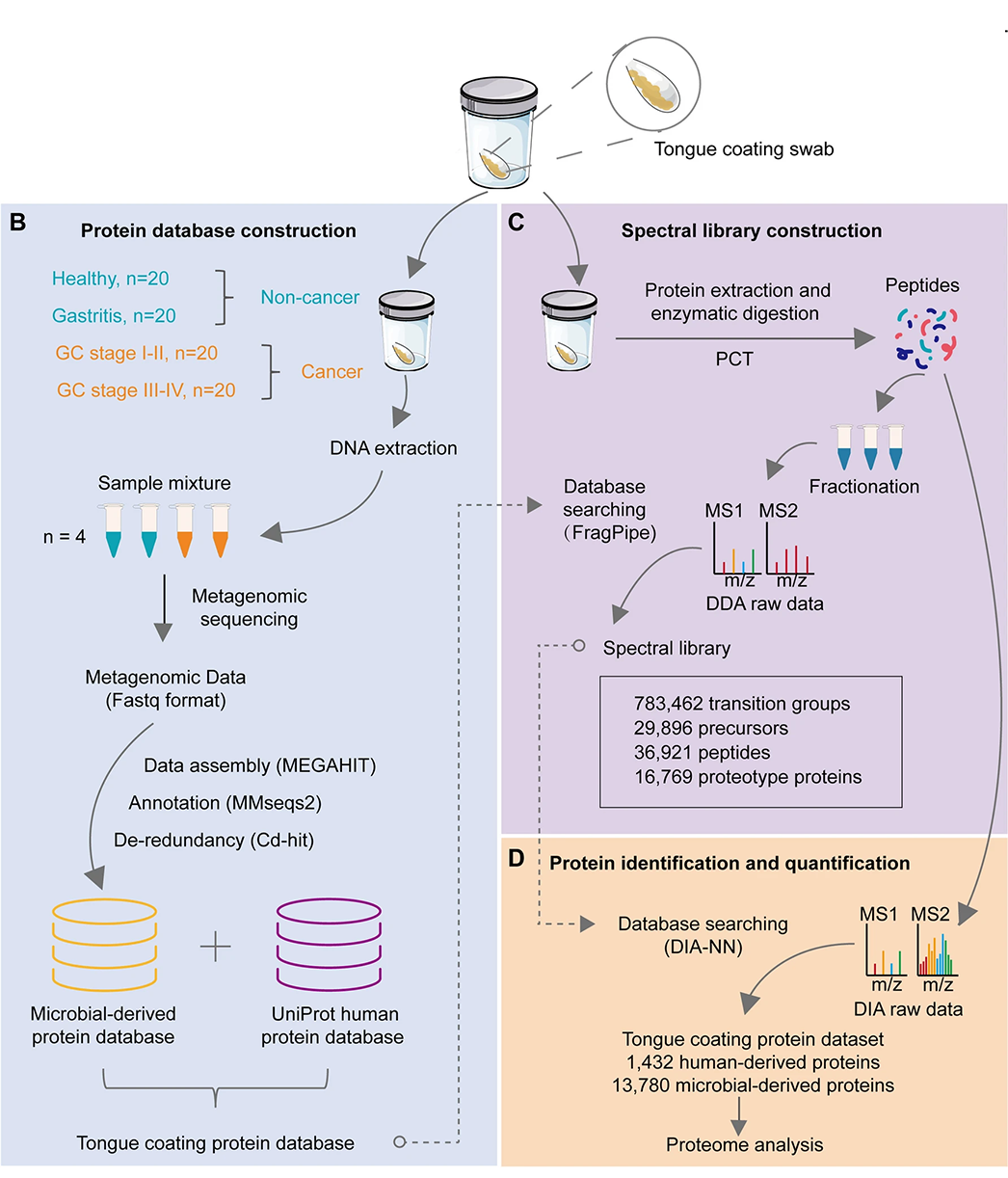
图3 构建舌苔蛋白序列库流程示意图
(2)舌苔蛋白质组随时间变化的稳定性分析
为了探究舌苔蛋白质组随时间变化的稳定性,研究挑选了5名健康志愿者于第0、3、6和9天分别采集样本,每次采集3份。由于一名志愿者因口腔出血未能参与最后一次采样,最终纳入该队列的样本总数为57例(图4A)。结果显示,共鉴定出12839个(90.19%)微生物来源蛋白和1396个(9.81%)人源蛋白(图4B)。研究发现,舌苔中人源蛋白与微生物来源蛋白的强度比随时间变化出现波动(图4C)。为探讨波动产生的原因,研究对个体间和个体内的蛋白质组数据进行了相关性分析。结果表明,随着时间的推移,人源蛋白在个体内和个体间的稳定性都较高。而微生物来源蛋白在个体内稳定性较高,但在不同个体之间稳定性较低。鉴于微生物来源蛋白的时间稳定性和个体之间的差异性,它们可以作为表征个体状态的可靠指标(图4D-E)。
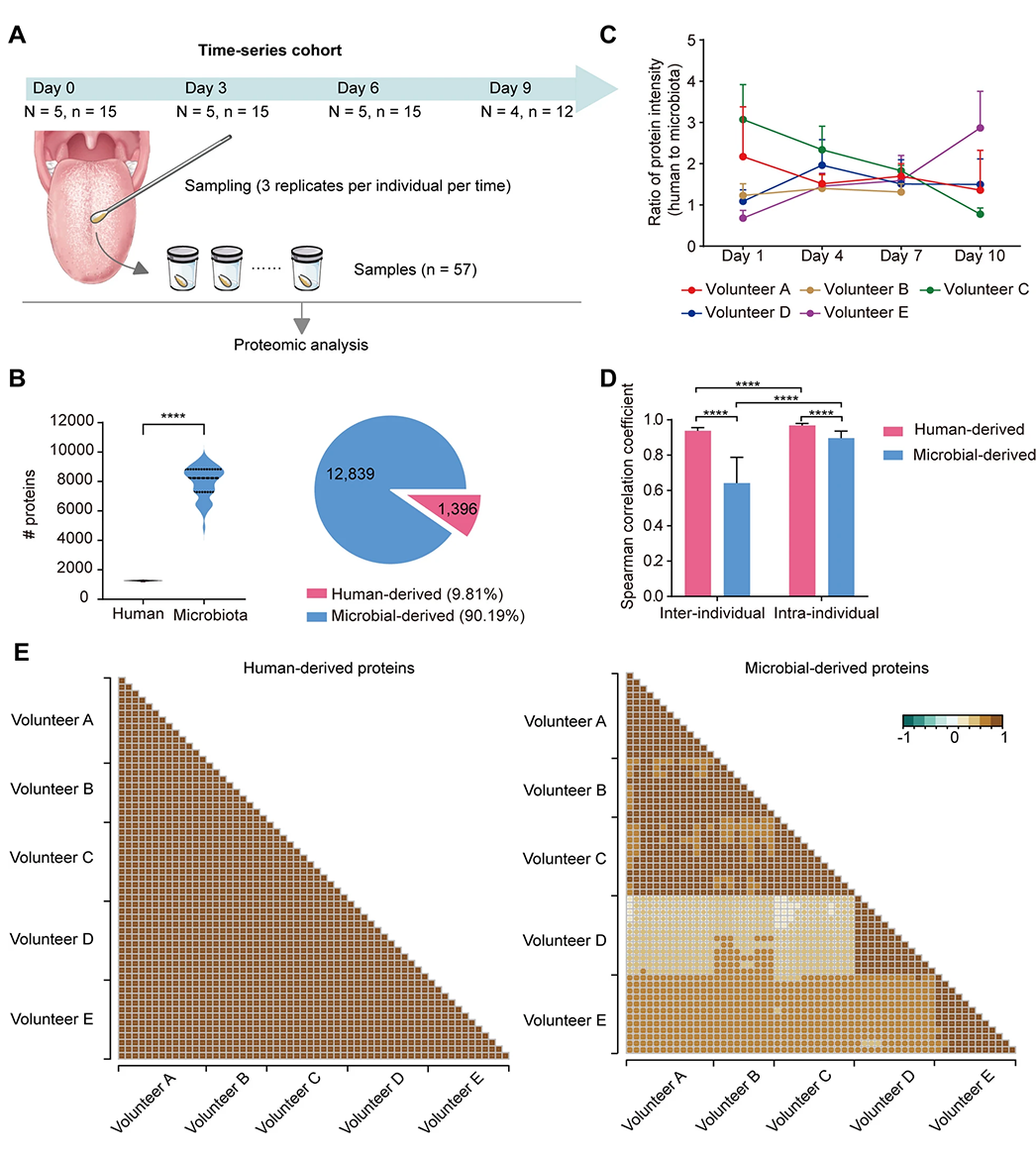 图4 舌苔蛋白质组随时间变化的稳定性分析
图4 舌苔蛋白质组随时间变化的稳定性分析
(3)舌苔蛋白质组的功能特征
接下来,研究比较了发现队列和验证队列的蛋白质组学数据。尽管这两个队列中的个体来自不同城市,但舌苔的蛋白质组学特征在两个队列中表现出较高的相似性(图5A-E)。其中,对微生物来源蛋白进行COG注释,结果显示共注释到1488个COG,其中96.5%在两个队列中都被鉴定到(图5A)。此外,为了探讨这些蛋白质在胃癌患者中的潜在生物学作用,研究还对发现队列中的人源蛋白和微生物来源蛋白进行了KEGG通路富集分析。结果表明,人源蛋白主要富集在细胞应激反应和中性粒细胞脱颗粒等通路(图5F),而微生物来源蛋白则主要富集于碳代谢和氨基酸生物合成等通路(图5G)。
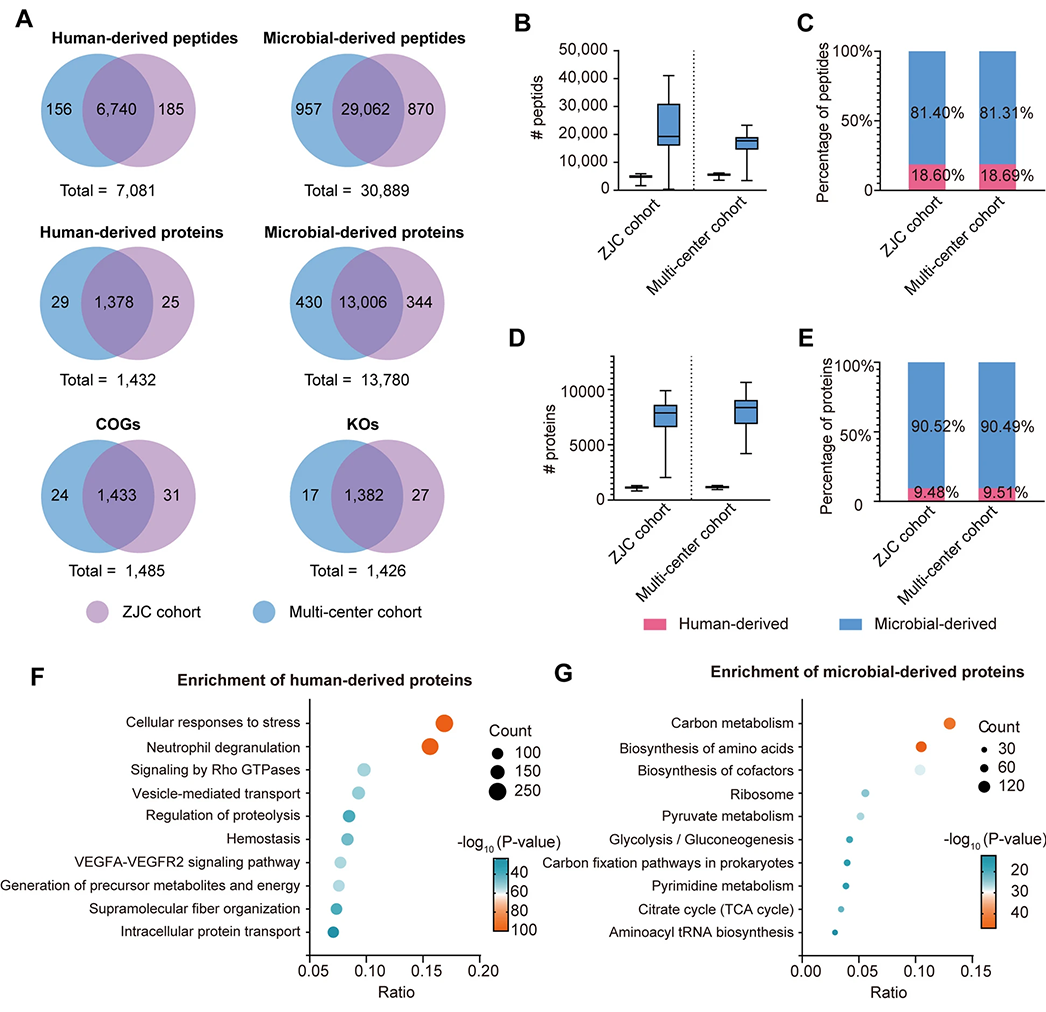
图5 舌苔蛋白质组的功能特征
(4)舌苔微生物群的分类
研究通过Unipept对所有舌苔样品中的微生物进行了注释,以初步了解舌头上细菌的多样性和数量。结果显示,舌苔中的微生物以细菌为主,而古细菌、真核生物和病毒的数量极少,仅各有1种。其中,发现队列的样本共鉴定出25个微生物门,其中以杆菌门、拟杆菌门、假单胞菌门、放线菌门和梭菌门为主(图6A)。同时研究比较了两个队列中的细菌,发现它们在门、纲、目、科、属和种水平上具有相似的舌苔微生物多样性(图6B-C)。
为了探讨舌苔微生物与胃癌发生的关联,研究进行了回归分析。结果显示,在物种水平上,一些细菌的丰度与胃癌风险呈正相关,而另一些则呈负相关(图6D)。遗憾的是,与胃癌发生显著相关的细菌种类在两个队列中的验证结果并不一致。
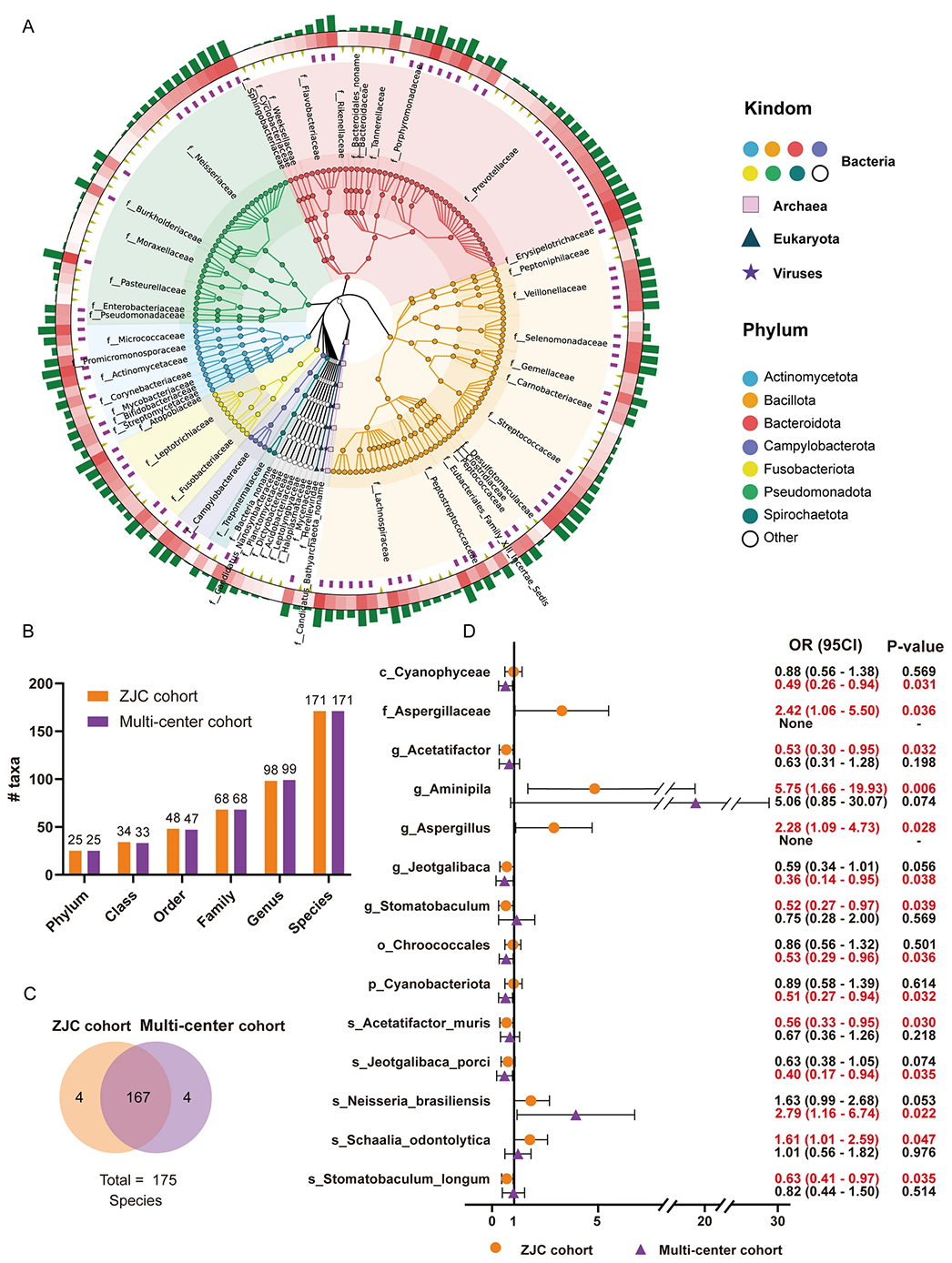
图6 舌苔微生物群的分类
(5)胃癌患者舌苔微生物群的功能变化
为深入探索胃癌患者舌苔蛋白的变化,研究对鉴定出的人源蛋白进行了差异表达分析。结果显示,在两个胃癌患者队列中,共同鉴定出了5种下调人源蛋白,包括KRT2、KRT9、DCD、EWSR1和CACNA1G,其中KRT2和KRT9是构成舌苔的重要角蛋白(图7A)。功能富集分析表明,下调人源蛋白主要与舌上皮的生长和分化相关,提示胃癌患者的舌表面物理屏障可能受到了削弱(图7B)。
为了探讨人源蛋白与微生物来源蛋白之间的相互关系,研究对KRT2、KRT9以及所有鉴定的COG蛋白的丰度进行了相关性分析。结果显示,COG1136与KRT2和KRT9呈高度正相关,且在胃癌患者的舌苔样本中,COG1136的表达水平明显低于非胃癌组(图7C-H)。COG1136是微生物ABC转运蛋白的一部分,该转运蛋白在细菌适应恶劣环境中发挥关键作用。研究推测,胃癌患者舌表面物理屏障的削弱可能降低了细菌的生存压力,从而导致ABC转运蛋白的表达水平下降。最后,研究对胃癌患者下调微生物来源蛋白进行了功能富集分析(图7I)。
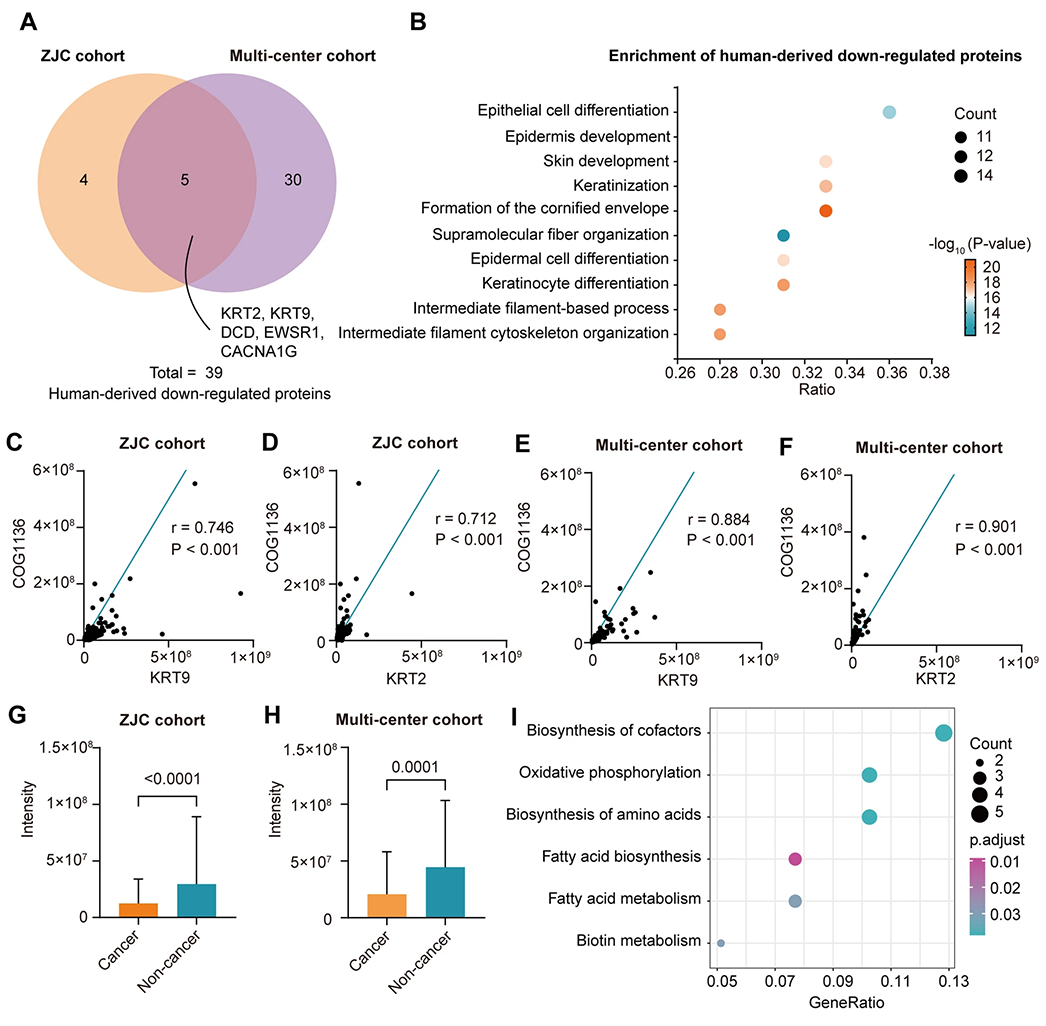
图7 胃癌患者舌苔微生物群的功能变化
(6)基于舌苔微生物蛋白建立的胃癌筛查模型
上述分析发现,微生物来源蛋白可作为可靠的个体表征指标来筛查胃癌患者。这使得基于舌苔微生物蛋白构建胃癌筛查模型成为可能。研究将发现队列人群按3:1的比例分为训练集和测试集,并使用随机梯度提升模型(GBM)筛选出训练集中最重要的50种微生物来源蛋白,进而建立胃癌筛查模型(图8A)。基于这50种微生物来源蛋白的t分布随机邻域嵌入(t-SNE)分析的结果显示,胃癌患者与非癌症个体能够被有效区分(图8B)。随后,研究使用ROC曲线图展示了该模型在训练集、测试集和验证队列中的表现,并用桑基图及一些常见指标展示了模型的性能(图8C-E)。结果表明,该模型具有较高的性能,舌苔微生物蛋白可以作为胃癌分类的有效生物标志物。最后,研究还对这50种微生物来源蛋白进行了功能富集分析(图8F)。
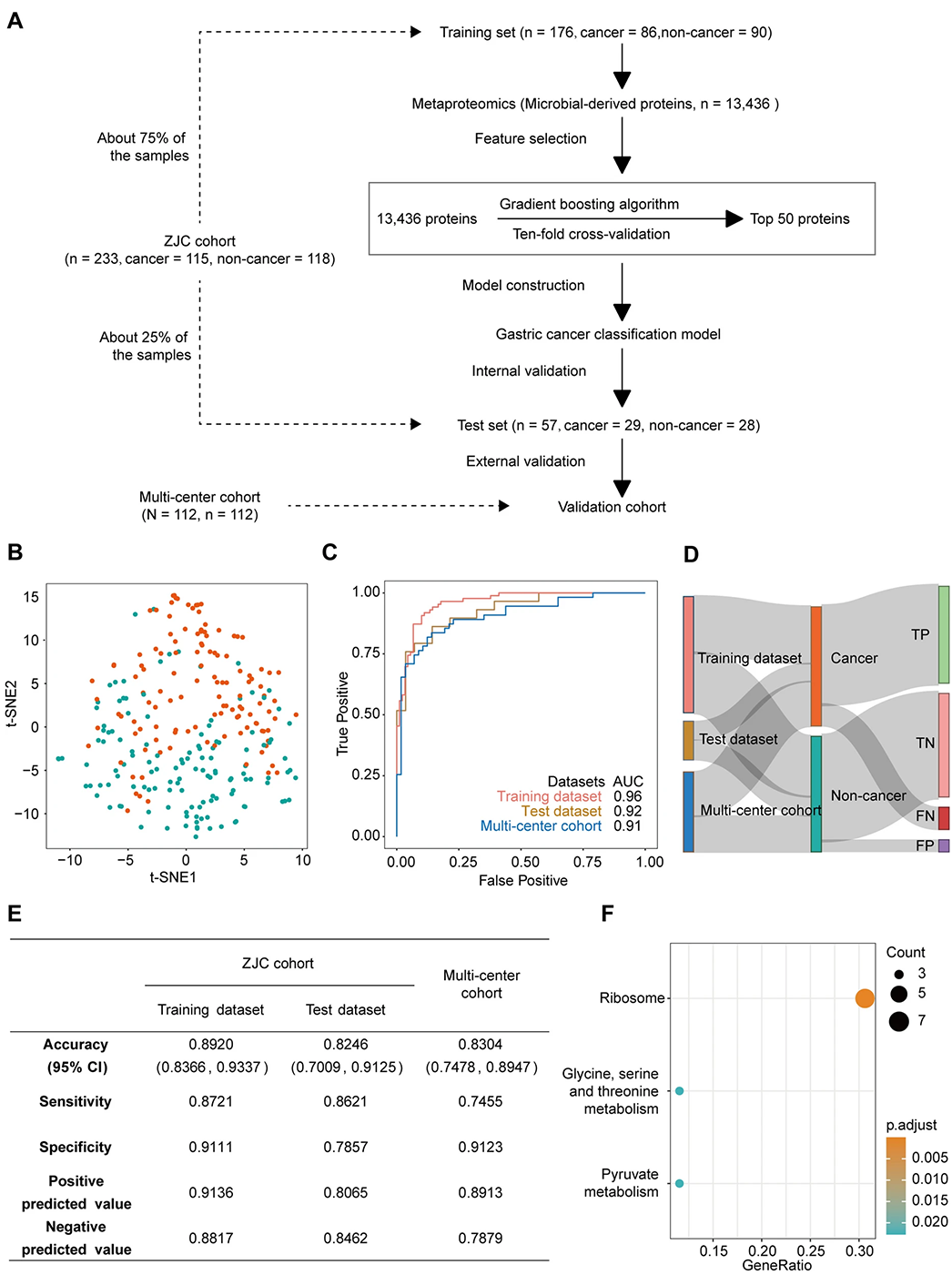
图8 基于微生物蛋白建立的胃癌筛查模型
♦结论
研究揭示了舌苔中人源蛋白与微生物来源蛋白的差异,并发现了一些与胃癌风险相关的微生物菌群。研究筛选并评估了具有重要临床意义的微生物来源蛋白,并通过机器学习的方法基于这些蛋白建立了胃癌筛查模型。该研究在寻找潜在胃癌无创生物标志物方面取得了重要进展,为胃癌的非侵入性筛查提供了新的方法。
♦总结
本研究是一项完整的诊断性研究,全篇未使用基础实验验证。作者建立了舌苔样本的特定谱库,简化了舌苔蛋白质组学的工作流程,并能扩展应用于更广泛的人群。同时证明了微生物来源蛋白在个体内具有良好的时间稳定性,个体间具有较大的差异性。又通过PCT-DIA蛋白质组学检测方法获得了具有临床价值的生物标志物,成功建立了舌苔微生物来源蛋白的高精度胃癌筛查模型。这两点的结合可以表明本研究开发了一种适合长期监测的潜在胃癌无创生物标志物。
但本文也存在一些局限性,比如在将舌苔蛋白作为生物标志物应用于大范围人群之前,还需进一步探讨饮食习惯和地理差异对其的影响。其次在复杂的临床环境中,不同疾病间存在大量重叠的微生物蛋白,区分疾病特异性变化仍然具有挑战性。
♦头脑风暴
本研究通过分析舌苔样本,利用蛋白质组学探讨了口腔微生物与胃癌的关系。看完这个案例,是否激发了大家的研究兴趣呢?我们可以借鉴该研究的设计思路,探索更多类似的研究方向。以下是小谱提出的几个研究方向,供大家参考:
a.唾液样本:口腔微生物与肺癌的关系
b.粪便样本:肠道微生物与抑郁症的关系
c.阴道分泌物样本:阴道微生物与早产的关系
通过深入解读本篇文献,相信大家对如何运用蛋白质组学技术进行临床研究有了更全面的理解。
以上就是今天的全部分享内容,如有任何疑问,欢迎随时联系!我们可为您提供全方位的蛋白质组学一站式解决方案,涵盖方案设计、报告生成以及协助论文发表等关键环节。我们致力于简化科研流程,为您提供全程科研服务,助您摆脱繁琐操作和耗时任务,为您的科研保驾护航!


















