
在上一期的分享中,我们探讨了如何利用TCMSP和SwissTargetPrediction数据库获取中药活性成分及其靶点信息。今天,我们将深入疾病研究和药物研发的核心领域——精准识别与疾病相关的靶点。这些“靶点基因”是连接中药活性成分与疾病治疗的关键纽带。OMIM(Online Mendelian Inheritance in Man)、GeneCards以及TDD(Therapeutic Target Database)等权威数据库为我们提供了海量的数据支持,帮助我们高效锁定疾病相关靶点。在今天的教程中,小谱将带领大家掌握如何利用这些数据库高效获取疾病靶点信息。
OMIM 数据库使用教程
OMIM全称叫做Online Mendelian Inheritance in Man,是人类在线孟德尔遗传数据库。它专注于人类基因和遗传疾病,详细记录了单基因疾病相关信息,包括疾病的临床描述、遗传模式、致病基因等,数据权威且更新及时,是研究遗传性疾病靶点的重要资源。
① 打开数据库:在浏览器地址栏输入网址:
https://omim.org/search/advanced/geneMap,进入OMIM geneMap页面。
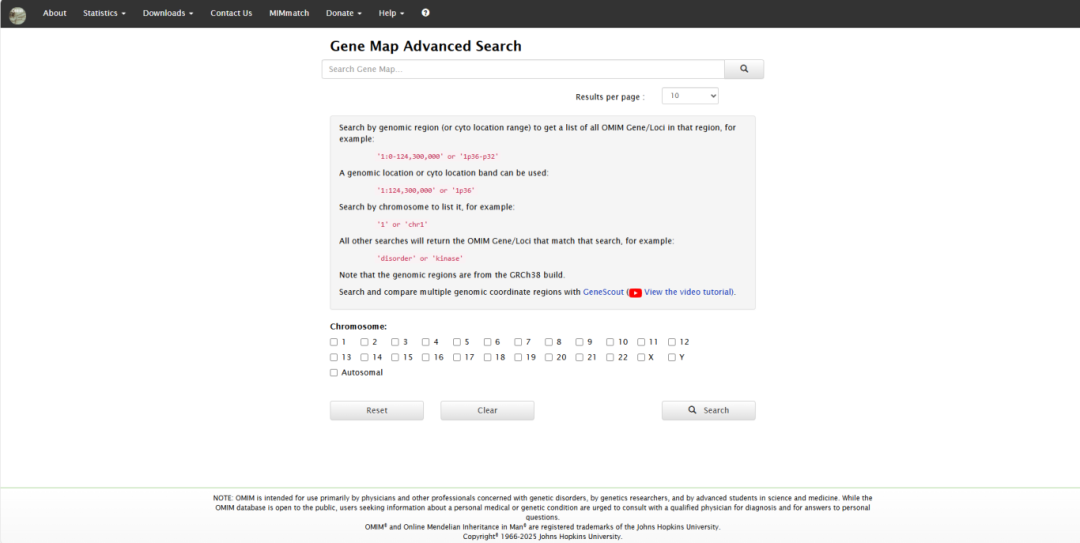
② 疾病搜索:在首页搜索框中输入目标疾病名称,例如“diabetes(糖尿病)”,点击搜索按钮(数据库支持使用疾病全称、缩写或相关症状等关键词进行搜索)。
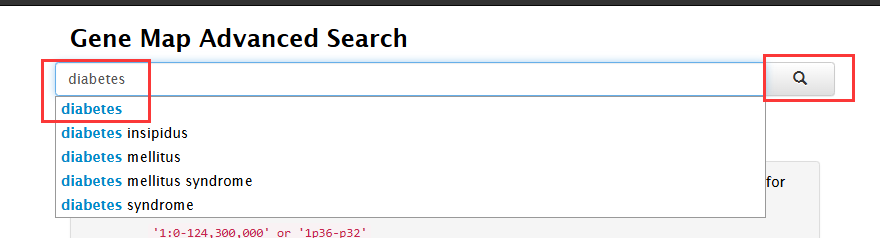
③ 获取靶点信息:搜索结果页面会列出与疾病相关的靶点基因。点击“Download As”项目下的“Excel File”,即可下载全部信息。
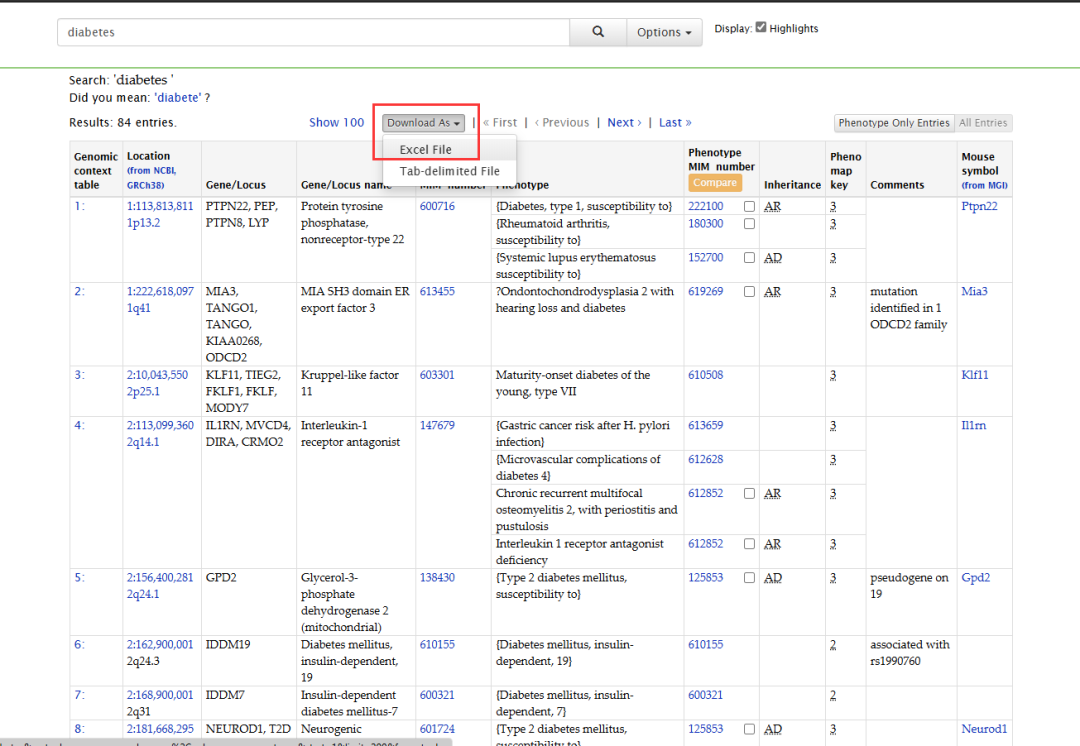
④ 数据整理:打开下载好的Excel文件,重点关注C列的“Gene/Locus”项。将此列靶点数据复制到Excel的新工作表中。
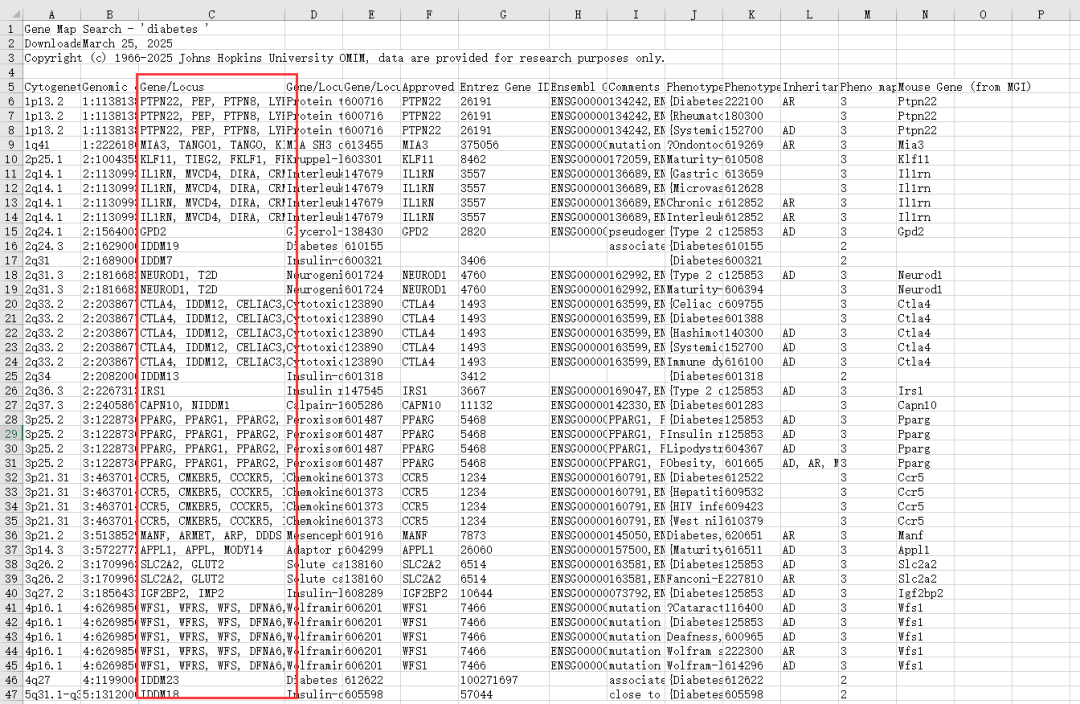
选中“Gene/Locus”列的所有数据,点击Excel的【数据】选项卡,选择【分列】功能。
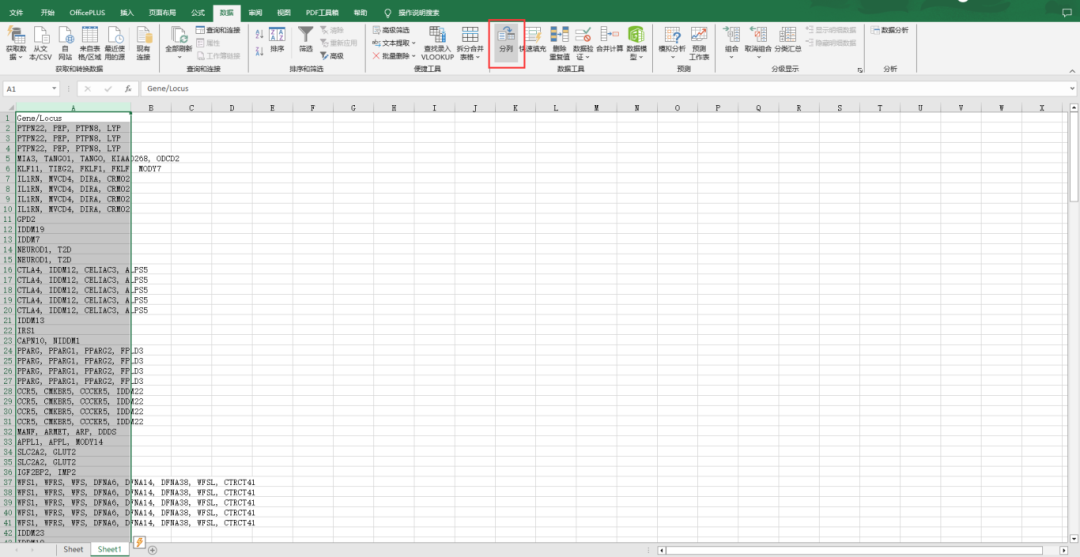
然后依次选择【分隔符号】→【逗号】→【常规】→【完成】,可以看到数据变成如下格式:
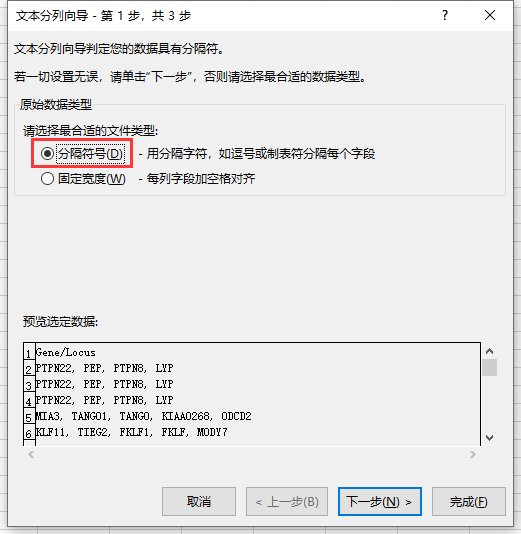
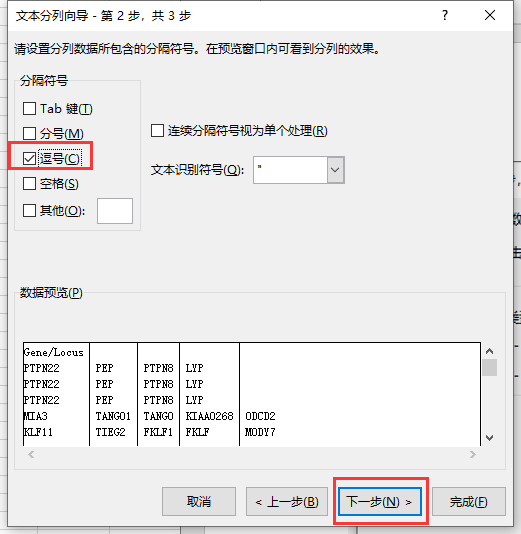
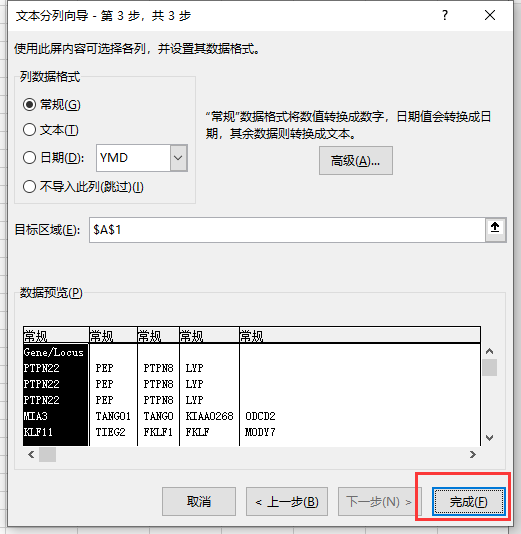
接下来,分别选中每列数据,点击【删除重复值】,并选择【以当前选定区域排序】。
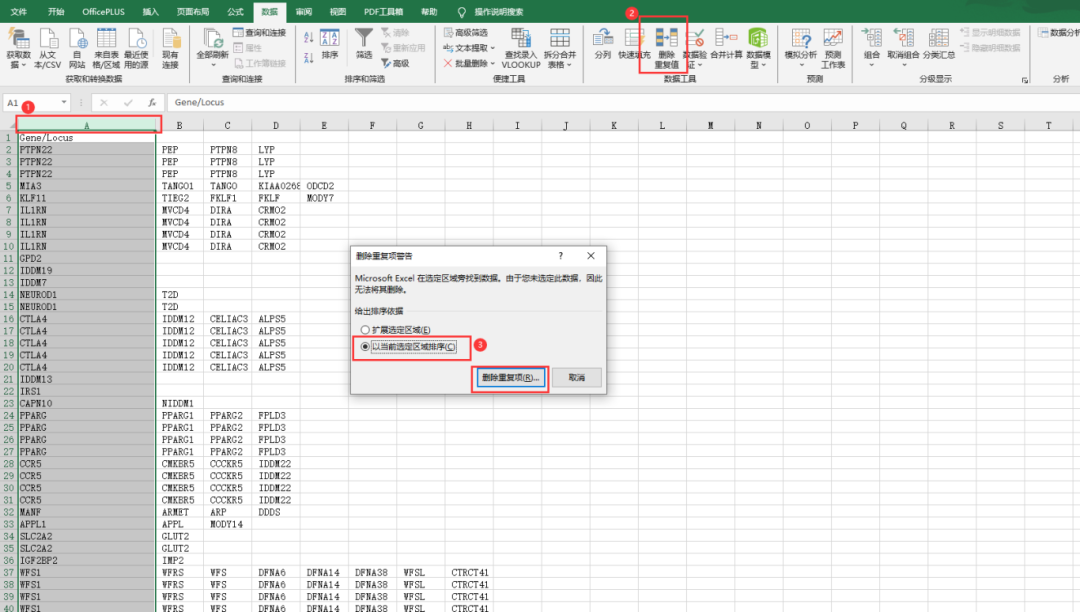
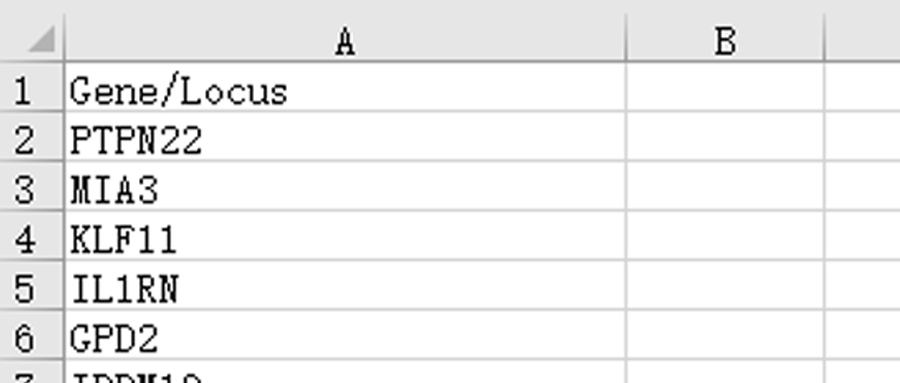
最后,将所有列的数据全部合并到一列,并再次【删除重复值】去重,这样,您就可以得到从OMIM数据库获取的糖尿病靶点表格文件了。
使用OMIM数据库时,推荐引用以下文献:
Hamosh A, Scott A F, Amberger J S, et al. Online Mendelian Inheritance in Man (OMIM), a knowledgebase of human genes and genetic disorders[J]. Nucleic acids research, 2005, 33(suppl_1): D514-D517.
GeneCards数据库使用教程
GeneCards是一个综合性人类基因数据库,整合了多源数据,涵盖基因功能、表达、疾病关联等丰富信息。其可视化界面设计精良,方便用户快速获取和分析基因相关数据,是疾病研究和药物研发的得力助手。
① 访问数据库:在浏览器中输入GeneCards网址:https://www.genecards.org/,进入首页。
② 疾病搜索:在搜索框中输入疾病名称,如“diabetes(糖尿病)”,点击搜索按钮。
③ 获取靶点信息:在搜索结果页面,点击“Export”按钮,选择“Export to Excel”下载CSV文件。
④ 数据整理: 打开下载的CSV文件,筛选“Relevance score”列,保留评分≥1.0的靶点(根据需求可调整阈值)。此时,“Gene Symbol”列即为糖尿病靶点基因名称.
使用GeneCards数据库时,建议引用以下文献:
Stelzer G, Rosen N, Plaschkes I, Zimmerman S, Twik M, Fishilevich S, Stein TI, Nudel R, Lieder I, Mazor Y, Kaplan S, Dahary D, Warshawsky D, Guan-Golan Y, Kohn A, Rappaport N, Safran M, Lancet D. The GeneCards Suite: From Gene Data Mining to Disease Genome Sequence Analyses. Curr Protoc Bioinformatics. 2016 Jun 20;54:1.30.1-1.30.33.
TDD 数据库使用教程
TDD:治疗靶点数据库(Therapeutic Target Database),专注收录经实验验证的药物靶点信息。它涵盖了靶点分子类型、疾病关联及药物研发阶段等内容,为药物研发人员提供了明确的潜在靶点信息,助力筛选药物和研发前景评估,推动创新药物的开发进程。
① 访问数据库:在浏览器中打开TDD官网:https://db.idrblab.net/ttd/,进入首页。
③ 获取靶点信息:该数据库数据量相对较少,搜索结果条目一般为几条到几十条。由于无法批量导出数据,您需要自行复制靶点基因名(通常位于括号内),并手动保存到Excel中。
使用TDD数据库时,建议引用以下文献:
Y. Zhou, Y. T. Zhang, D. H. Zhao, X. Y. Yu, X. Y. Shen, Y. Zhou, S. S. Wang, Y. Q. Qiu*, Y. Z. Chen*, F. Zhu*. TTD: Therapeutic Target Database describing target druggability information. Nucleic Acids Research. 52(D1): 1465-1477 (2024).
数据整合与韦恩图绘制
将OMIM 、GeneCards、TTD等数据库获得的靶点汇总到同一个Excel表格中。使用Excel的【删除重复项】功能,生成去重后的靶点列表。最终的疾病靶点汇总表格应包含疾病名称、数据库来源、靶点名称等关键信息,确保数据清晰、有条理。
绘制韦恩图
使用在线工具(例如Venny2.1)绘制韦恩图,直观展示不同数据库中靶点的交集情况。如果某个靶点在多个数据库中均被识别为疾病相关靶点,那么该基因作为疾病靶点的可靠性将显著增强,可被视为关键靶点。这种方法不仅提高了靶点识别的准确性,还为后续研究提供了重要的参考依据。
下一步:构建“中药-成分-靶点-疾病”网络
按照本次分享的详细教程和步骤操作,相信大家都能从 OMIM、GeneCards和TTD数据库中高效获取感兴趣的疾病相关靶点。接下来,把这些疾病靶点和之前获取的药物靶点进行联合分析,并借助Cytoscape等网络分析工具,构建 “中药-成分-靶点-疾病”网络,就能直观呈现中药成分通过多靶点、多途径发挥作用的机制。关于构建该网络的具体方法,我们将在后续文章中详细介绍。
常见问题解答
以上就是我们今天的分享内容,在实际使用中,我们可能还会遇到以下疑问:
Q:GeneCards数据库中的靶点数量众多,并且会根据相关性评分(Relevance score),筛选时是否都依据评分达≥1.0的标准?
A:筛选条件在文献中有所差异,常见的标准包括:①Relevance score≥1.0;②Relevance score≥中位数;③Relevance score≥10.0,④直接不筛。实际上,并没有统一的筛选标准。研究者可以根据靶点的数量和研究需求自行调整筛选阈值。
Q:疾病靶点的来源数据库各不相同,在进行网络药理学研究时,是否需要检索所有这些数据库?
A:文献中通常建议至少从两个数据库中获取疾病靶点并进行汇总。当然,选择多个数据库进行检索也是可行的。研究者可以根据具体的疾病靶点情况,灵活选择两个或两个以上的数据库来获取疾病靶点信息。
Q:如果某些疾病在上述数据库中找不到靶点,该怎么办?
A:首先,可以尝试使用多个数据库进行搜索。此外,还有其他数据库可以提供疾病靶点信息,例如DisGeNET、DrugBank和Pharmgkb等。以下是这些数据库的链接:
DisGeNET数据库:https://disgenet.com/
DrugBank数据库:https://go.drugbank.com/
Pharmgkb数据库:https://www.pharmgkb.org/


















